Imaginez si nous pouvions suivre chaque cellule d’un organisme en développement. Le volume d’information, sur la façon dont se forment et fonctionnent les organismes biologiques, serait précieux pour la recherche scientifique. Le seul problème, c’est la puissance de calcul qu’il faudrait pour assimiler un énorme volume de données contenues dans les observations au microscope. Une équipe de scientifique a réussi à contourner cela.
Petit retour en arrière…
En 2012, Philipp Keller du Howard Hughes Medical Institute (Janelia Farm) a développé le simultaneous multi-view (SiMView) Light sheet fluorescence microscopy, une technique de microscopie à fluorescence qui capture des images en trois dimensions avec une vitesse et une précision sans précédent, sur des heures ou des jours. En 2013, la même équipe a été en mesure d’enregistrer les sursauts de l’activité cérébrale chez un poisson, toutes les 1,3 seconde, affichant 80 % de ses 100 000 neurones.
Aujourd’hui, ils annoncent la création de ce qui ne peut être décrit comme un super logiciel pour visualiser l’univers cellulaire en expansion…
Ils ont développé une nouvelle méthode de calcul qui peut rapidement suivre les mouvements en trois dimensions des cellules dans leurs images riches en données. En utilisant cette méthode, les scientifiques peuvent automatiser une grande partie du processus consommant le plus de temps dans la reconstruction, cellule par cellule, du “plan” de développement d’un animal.
Le programme permet ainsi de suivre et de rendre les mouvements tridimensionnels des cellules au cours du développement. Il peut également accélérer ou inverser le processus afin d’acquérir de nouvelles idées sur la façon dont ces sphères de matière organique s’organisent dans les beaux systèmes du vivant. En plus, le logiciel (open source) est suffisamment efficace pour fonctionner sur la carte graphique d’un ordinateur de bureau moyen (vous trouverez le lien pour le télécharger en bas de cette page).
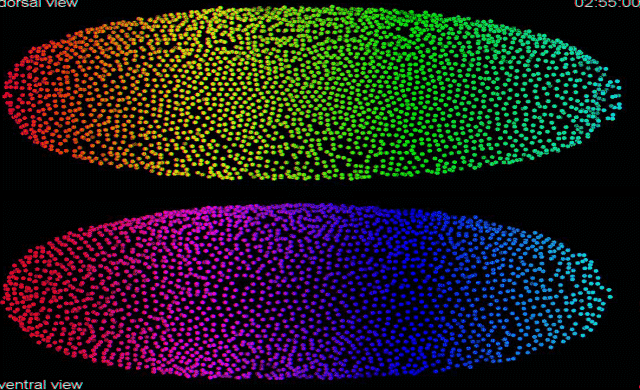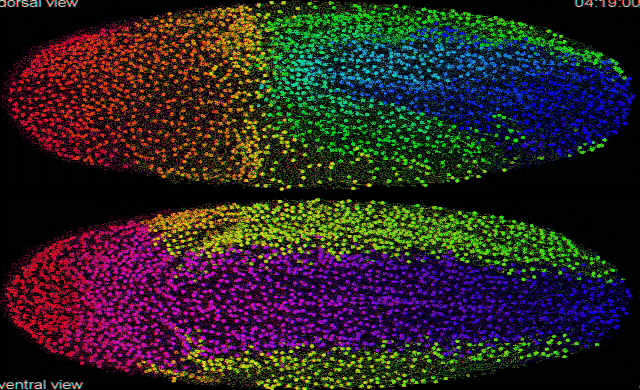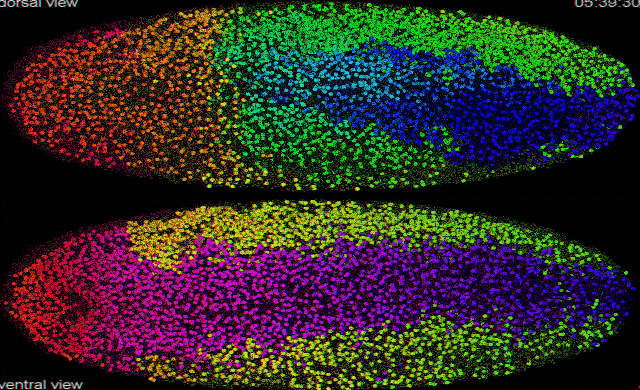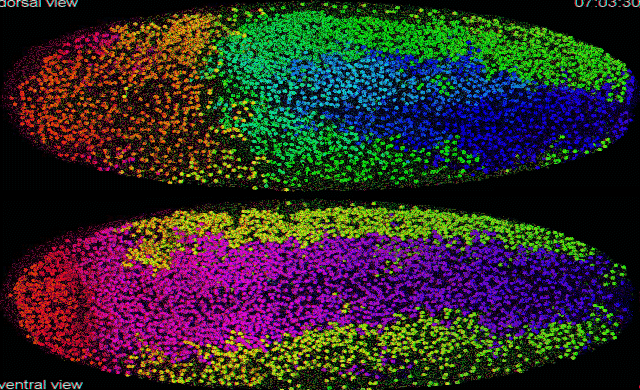
Sur le GIF d’entête : “Segmentation automatique et le suivi des données SiMView de l’embryogenèse d’une mouche drosophile” : Cette vidéo présente le calcul automatisé de données image pour produire la reconstruction cellulaire du développement d’un embryon de drosophile. Chaque cercle représente un noyau cellulaire.
L’immense espoir, pour cette nouvelle technique d’imagerie cellulaire, est d’être en mesure de mieux comprendre comment les choses tournent mal et comment se développent les maladies héréditaires.
D’autres reconstructions consultables dans la partie vidéo ici et l’étude publiée dans Nature Methods : Fast, accurate reconstruction of cell lineages from large-scale fluorescence microscopy data
Les scientifiques détails leurs travaux sur le site de la Janelia Farm : Keller Lab ou vous trouverez une partie “Software” en bas de page afin de télécharger l’application.