Bon, vous l’aurez sans doute remarqué, la nature ne s’embête avec des éléments inutiles, de par son évolution, elle s’optimise continuellement et les rares éléments biologiques du corps humain dont on ne trouve une quelconque utilités, sont les empreintes d’un passé qui est encore entrain de s’effacer et heureusement… Il en va de même pour cette nouvelle carte du génome humain, qui permet de constater que des éléments de régulations génétiques représentent 80 % de notre ADN.
De longues chaines de l’ADN, précédemment qualifiées de “poubelle” (ADN non codant, “Junk ADN” en anglais) sont en fait cruciaux dans la manière dont fonctionne notre génome, selon la déclaration d’une équipe internationale de chercheurs cette semaine.
Seulement 1 %, environ, du génome humain contient des régions de gènes qui codent les protéines, ce qui soulève la question de ce que le reste de l’ADN fait.
Les scientifiques ont maintenant commencé à découvrir la réponse et selon l’équipe internationale de chercheurs qui a publié cette nouvelle étude (lien plus bas): environ 80 % du génome est biochimiquement actif et probablement impliqué dans la régulation de l’expression des gènes voisins. 
Le consortium, connu sous le nom d’ENCODE (Encyclopedia of DNA Elements), comprend des centaines de scientifiques de plusieurs dizaines de laboratoires à travers le monde. En utilisant les données du séquençage génétique de 140 types de cellules, les chercheurs ont pu identifier des milliers de régions d’ADN qui permettent d’affiner l’activité des gènes et d’influencer ceux qui sont exprimés dans différents types de cellules.
Tout comme le séquençage du génome humain a aidé les scientifiques à comprendre comment les mutations des gènes codant des protéines peuvent provoquer la maladie, la nouvelle carte des régions non codantes devrait apporter des réponses sur la façon dont les mutations, dans les éléments de régulation, conduisent à des maladies telles que le lupus et le diabète, explique Manolis Kellis, professeur associé d’informatique au MIT (Massachusetts Institute of Technology), membre associé de la Broad Institute et auteur d’un document décrivant les résultats dans un article en accès libre le 5 septembre (lien plus bas).
Kellis précise :
Les êtres humains sont à 99,9 % identiques entre eux et vous n’avez qu’une seule différence dans tous les 300 à 1000 nucléotides. ENCODE nous permet de fournir une annotation de ce que fait chaque nucléotide du génome, ainsi, quand il est muté, on peut établir des prédictions sur les conséquences de la mutation.
Kellis, qui dirige le Groupe MIT Computational Biology, est l’un des principaux chercheurs impliqués dans la recherche. La collaboration ENCODE est encore en train de publier environ deux douzaines d’articles supplémentaires cette semaine, qui détaillent les nouveaux résultats.
La cartographie de l’ADN non codant
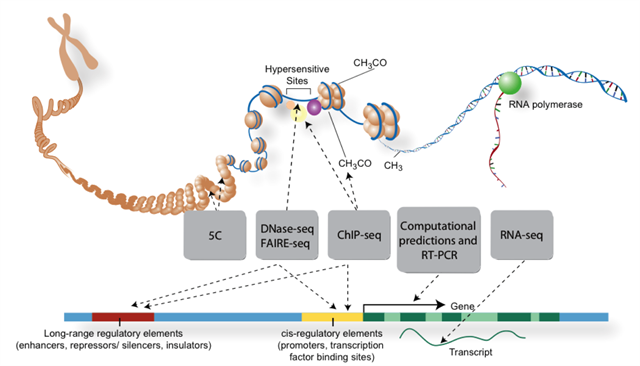
ENCODE a été créé en 2003, pour étendre notre compréhension du génome humain au-delà de gènes codant les protéines. Pour ce faire, il faut étudier les modifications chimiques de chaque tronçon de l’ADN, qui contrôlent la mise en activité de ces régions génétiques. Ces modifications varient selon le type de cellules et peut modifier directement, à la fois l’ADN ou les protéines histones sur lesquelles s’enroulent l’ADN.
Pour mapper ces modifications, connues collectivement sous le terme d’épigénome (modification épigénétique), les groupes de recherche ont dû rassembler de nombreux types de données provenant de différents types de cellules. Certains laboratoires ont mesuré les modifications ADN ou des histones, tandis que d’autres ont jaugé l’accessibilité des différents tronçons de l’ADN en les coupant en morceaux avec des enzymes.
Le Guru a déjà écrit sur le sujet épigénétique : Les gènes sont-ils notre unique destinée ? Quelque chose de plus rapide se cache derrière notre code génétique.
Kellis et son groupe ont été parmi les principaux scientifiques à analyser et intégrer l’énorme quantité de données générées par les différents laboratoires.
Anshul Kundaje, un chercheur du MIT dans le groupe Computational Biology, explique :
Étant donné que nous recevions plus de 1 000 groupes de données, nous avons dû trouver des façons de calibrer automatiquement les expériences. Nous avons développé un système presque entièrement automatique qui fait tout cela.
Les chercheurs d’ENCODE ont constaté que 80 % du génome subit une sorte d’évènement biochimique, comme la liaison aux protéines qui régulent la fréquence à laquelle un gène voisin est utilisé. Ils ont également découvert que la même région de régulation peut jouer différents rôles, selon le type de cellule.
Les résultats devraient avoir un impact majeur sur la compréhension, par les scientifiques, de la biologie humaine et de la façon dont les variations génomiques peuvent causer des maladies.
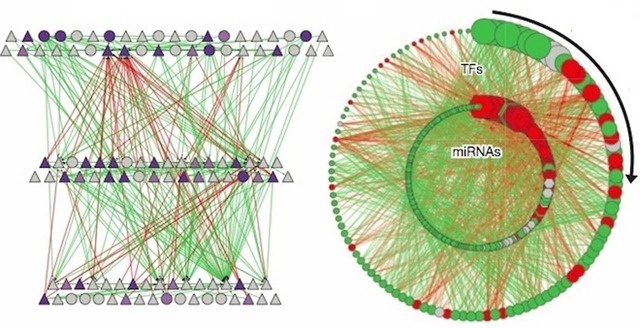
Ci-dessous : visualisations des liaisons interconnectées entre les composantes génétiques à travers le génome humain (à droite) et en un plus petit sous-ensemble arrangé hiérarchiquement (à gauche). (Gerstein et al./Nature)
La variation humaine
Les chercheurs ont également étudié la conservation de nucléotides, les “lettres” C A, T, G de l’ADN, dans les régions de régulations nouvellement identifiées. Les nucléotides sont conservés si elles restent les mêmes au cours de longues périodes évolutives qui peuvent être mesurées par l’analyse de la variabilité entre les espèces ou entre les individus d’une même espèce.
Un article récent de Kellis et ses collègues a montré que 5 % de l’ADN non codant est conservé à travers les mammifères. Dans l’un des documents complémentaires d’ENCODE, apparaissant en ligne le 5 septembre, Kellis et Lucas Ward du MIT montrent qu’un supplément de 4 % est conservé au sein de la lignée humaine, ce qui suggère que ces éléments contrôlent des traits qui ont récemment évolué, dont certains sont uniques à l’homme.
Lorsque les chercheurs ont étudié les fonctions des gènes, qui ont récemment évolué près des régions régulatrices, ils ont trouvé de nombreux gènes qui codent les régulateurs qui activent d’autres gènes. Les gènes impliqués dans la croissance neuronale et la vision des couleurs, qui ont tous deux été supposés être des innovations récentes chez les primates, sont enrichis en éléments humains limités dans les régions non conservées.
Les chercheurs ont constaté que les nucléotides les plus conservées sont aussi les plus susceptibles d’être associés à la maladie lorsqu’ils sont mutés. Ils ont également montré que des variantes associées à des maladies auto-immunes, telles que le lupus et l’arthrite rhumatoïde, sont uniquement situées dans les régions actives des cellules immunitaires, tandis que les variantes liées à des maladies métaboliques sont uniquement dans les régions actives des cellules hépatiques.
Dans la vidéo ci-dessous, le principal coordinateur d’ENCODE, Ewan Birney et le rédacteur en chef de la revue Nature, Magdalena Skipper, parlent des défis pour diriger ce projet colossal et de ce que nous avons appris de notre génomes.
Dans la phase suivante, les chercheurs d’ENCODE espèrent déterminer à quel point ces variations conduisent à une maladie humaine.
Selon Manolis Kellis :
Ce que nous avons fait, au cours de cette série de documents, est de dépeindre un ensemble d’annotations de référence concernant la fonction du génome humain commun. Nos prochaines étapes seront de personnaliser ces cartes, de se demander comment elles varient naturellement entre les individus, par le profilage de différents types de cellules de différentes personnes et comment leur variation concerne les maladies humaines et les traits humains complexes.
Dans un autre des projet, qui va en découler, Kellis et ses collègues comparent les niveaux d’activité des éléments de régulation dans différents types cellulaires chez une même personne, à travers de nombreux individus. Un autre projet est à la recherche des modèles de modification d’ADN dans le génome de nombreuses personnes, dans l’espoir d’identifier comment la variation d’éléments spécifiques se rapporte à une maladie.
Image d’entête : les chercheurs d’ENCODE emploient une variété de tests et de méthodes pour identifier les éléments fonctionnels. La découverte et l’annotation des éléments du gène est principalement accomplie par le séquençage ARN d’une gamme variée de sources, de comparatifs génomique, de méthodes bio-informatique intégratives et humaine. Les éléments de régulation sont généralement étudiés grâce à des essais d’hypersensibilité de l’ADN, des essais de de méthylation de l’ADN et d’immunoprécipitation de la chromatine (ChIP) des protéines qui interagissent avec l’ADN, y compris les histones modifiées et les facteurs de transcription, suivis par le séquençage (ChIP-Seq).
La recherche a été financée par le National Human Genome Research Institute.
Le site ENCODE : Encyclopedia of DNA Elements. L’annonce sur la Broad Institue : Mapping a genetic world beyond genes.
L’étude en libre accès sur Nature : An integrated encyclopedia of DNA elements in the human genome.


99,9 % seulement de similarité génétique ?
J’aurais pensé bien plus. Vu qu’on doit bien être à 99,99999 si on ajoute l’absence de diversité représentationnelle, idéologique ou religieuse.
Comme si la culture consolidait l’homogénéité, finalement assez relative, de la nature, au lieu de faire l’inverse comme nous sommes tentés de le penser.
Cela veut dire que notre ADN est le résultat d’un code réfléchi comme un code informatique … il y aurait donc un programmeur ? comme a dit Voltaire “L’univers m’embarrasse, et je ne puis songer que cette horloge existe et n’ait pas d’horloger.” nous ne devons pas oublier que nous sommes des créatures et qu’il y a un créateur qui nous aime personnellement.